GBS技术简介:
GBS(Genotyping-by-Sequencing)技术指通过测序进行基因分型,通过选取合适的限制性内切酶结合高通量群体测序构建SNP分子标记,可用于分子标记开发、超高密度遗传图谱构建、群体遗传分析、群体GWAS分析等。
实验技术新升级:
1. 诺禾为您提供个性化的实验方案,我们的内切酶库有MseI,EcoRI,NlaIII,HaeIII等十几种限制性内切酶,可根据不同物种、不同科研目的,通过生物信息学评估为您选择最为合适的限制性内切酶。
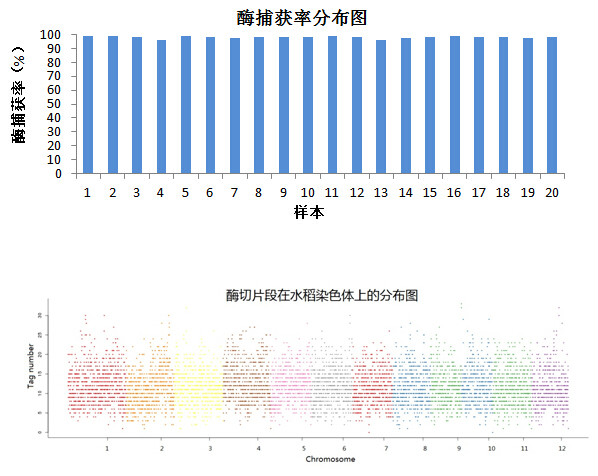
2. 诺禾测序工厂使用原装进口试剂,保证测序质量Q30在85%以上,酶切捕获效率达95%以上,且捕获的酶切片段在染色体上分布的均一性好。
3. 诺禾采用升级后的PE125测序读长,对基因组的覆盖度更高,捕获数据量更大,获得的信息量相对于其他简化基因组技术提升了26%。
生物信息分析流程新升级:
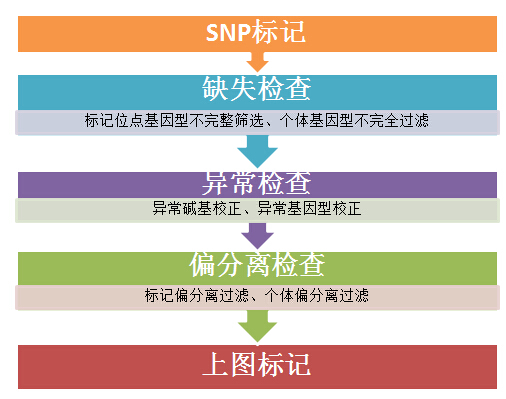
除常规信息分析手段外,诺禾自主研发了多项生物信息分析流程,对获得的SNP标记,进行缺失检查、异常检查、偏分离检查,最终保证高质量的上图标记。
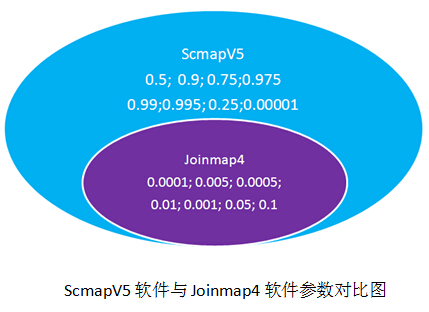
诺禾自主研发的ScmapV5构建遗传图谱软件可完美匹配高通量测序数据,使遗传重组率计算更准确,图距更真实,并已发表高影响因子文章(IF: 31.477),得到行业内认可。
案例分享:
利用GBS技术构建玉米遗传图谱和QTL定位研究(BMC Genomics,2014)
通过对710份玉米单株进行GBS建库测序,获得1,155,158个SNP,构建了一个含有6533个bin-makers的遗传图谱,遗传图距长度为1396 cM。bin-makers之间的遗传图距从0.1 cM到11.5 cM,平均遗传图距为0.2 cM。通过对2个已知基因定位,确认了该图谱的高质量和高准确性。在F2中,评估了雄穗分枝数、穗行数及雌穗长度三个性状,得到10个QTL,其中7个与前人报道的QTL相重叠。三个MADS-box蛋白和一个BTB/POZ蛋白编码基因位于qTBN5和qTBN7(分别长为800Kb与1.6Mb)之中,可能参与了雄穗构造的形成。

参考文献
An ultra-high density bin-map for rapid QTL mapping for tassel and eararchitecture in a large F2 maize population. BMC Genomics,2014,15:433.







