
戴俊彪
本文转载自“澎湃新闻”。
这些研究工作均为国际科研项目“人工合成酵母基因组计划(Sc2.0)”的一部分,该项目由美国科学院院士Jef Boeke主导。一年之前,《科学》杂志也在同一天在线发表了国际团队共同完成的7篇专刊文章。
中国科学院深圳先进技术研究院合成生物学所合成基因组学研究中心的领头人戴俊彪是Sc2.0项目的主要负责人之一。在去年的那批成果中,戴俊彪及其团队攻克的是酵母中最长的那条染色体(12号合成染色体)的人工合成。此次发布的最新进展中,戴俊彪课题组和英国曼彻斯特大学蔡毅之课题组合作发表了其中2篇论文。
戴俊彪在接受澎湃新闻(www.thepaper.cn)采访时表示,“要想用这个SCRaMbLE系统,实际上最好需要一套严格的筛选系统,把染色体上发生了重组的细胞筛选出来,所以我们就开发了ReSCuES系统,帮助我们非常有效地筛选基因组发生了重排的菌株。”
而所谓的SCRaMbLE系统,则是贯穿了此次7篇论文的一个“主角”。 该系统可以在短时间内快速地让合成的酵母染色体发生重新排列,被认为赋予了进化“超能力”。而在大自然演化中,物种的基因组序列在整个生命过程中通常需要维持相对稳定,以保证生命机器的有序进行,现如今多样的基因组其实是经历了亿万年的循序渐化。
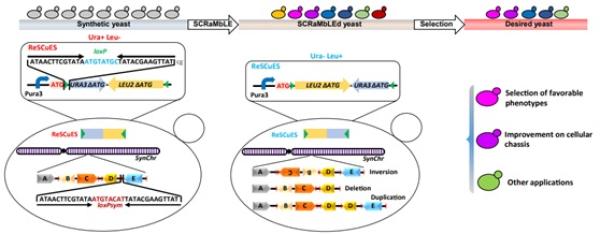
合成酵母的“进化加速器”——ReSCuES系统
给SCRaMbLE系统加道“保险”
SCRaMbLE系统最早于2011年由Boeke及其团队首次发表于《自然》。在当时的研究中,携带部分人工合成染色体(插入43个LoxPsym位点)的菌株在重组酶激活后最终产生了突变菌株,这些突变株在生长率方面表现出了很大的变化。
戴俊彪表示,“实际上,在设计合成染色体的时候,我们在每一个非必需基因的后面都掺入了一个LoxPsym位点,LoxPsym位点能被Cre重组酶识别。LoxPsym位点就相当于是特殊的标签,Cre重组酶进去之后抓出2个LoxPsym位点就能对对它们进行重组。“
也就是通过系统性地插入了大量的序列特异性重组位点loxPsym,成功在合成的酵母基因组中植入了短时间内从一个基因组出发,进化获得高度多样的基因组序列的“超能力”。 基因组序列发生了由“1”到“N”的变化。
戴俊彪和蔡毅之合作团队的工作则是确保合成染色体的基因组序列发生了变化,从而进一步为进化“提速”。
戴俊彪表示,细胞内的野生型loxP位点和loxPsym位点具有严格的正交性,即不会发生交叉反应,但是都能够被Cre重组酶识别并介导反应。利用这一特性,研究团队构建了一个正交性的报告系统ReSCuES,可以从SCRaMbLE后的混乱群体中精确筛选出发生基因组重排的细胞。
戴俊彪进一步解释,“筛选的标准是利用了2个标记,这2个标记在Cre重组酶条件下会翻转过来。例如一开始标记a是开着的,标记b是关着的;一旦重组以后,a就会变成关着的,b变成开着的。”
经ReSCuES筛选出来的每个菌株都含有其特异的基因组序列组成,这些菌株可作为原材料供后续筛选优良性状、构筑底盘细胞以及其他用途。
“SCRaMbLE、ReSCuES以及后续的筛选过程组成了一个高效的‘进化加速器’,可以在短短几天的时间内实现自然界中需要漫长的时间(可长达上亿年)才能完成的性状进化过程。”蔡毅之表示。
助力酵母菌株工业应用
值得一提是,与实验室培养不同,在工业应用过程中酵母通常需要面对来自环境中不同的胁迫,包括酵母自身发酵产生的乙醇、乙酸等有毒害作用的物质,以及来自发酵培养过程中的高温、高盐等不适的环境条件。
如何提高酵母对环境胁迫的耐受性一直是工业应用领域内的研究热点。
基于该“进化加速器”,研究团队首先以含有第12号合成染色体的菌株为模型,成功实现了乙醇耐受性的提升并提高了乙醇的发酵产量。然后以筛选获得的耐受菌株为对象,解析耐受性背后的机制为,3’非编码区调节Ace2p转录因子水平进而调控细胞乙醇耐受性。
最后研究团队以多个不同的合成菌株为起点,成功实现了高温耐受性和乙酸耐受性的提升。进化后的菌株在高浓度的乙酸条件下的生物量积累可以提高将近40倍。
除了提高菌株对不利因素的耐受性,快速实现目标产物代谢途径和底盘细胞的优化适配是目标产物产量最大化的另外一个重要方面。
形象来说,就如水利输送系统,为了达到最优的输送効率,不仅需要优化各步骤的运输能力,使它们相互协调(目标产物代谢途径优化),还需要增加来源和减少运输过程中的不必要的消耗(底盘细胞优化适配)。
针对这一问题,戴俊彪表示,“我们先在体外用SCRaMbLE的办法把一条代谢通路上的基因、调控元件进行重排,再把重排后的元件插入到合成酵母的基因组里面去。”经过这一步,实现了底盘基因组的多样化。随后,通过合适的筛选,即可获得外源代谢途径与底盘相互优化适配的高产菌株。
研究团队用提高β-胡萝卜素和紫色杆菌素产量验证了这一流程的高效。研究团队通过SCRaMbLE-in技术将优化后的代谢途径插入合成基因组中,并同时重排底盘细胞基因组,实现目标产物产量的第一次提升,即提升将近5倍。最后,通过多轮的SCRaMbLE实现生产菌株的产量的连续提升,最终产量为初始产量的将近20倍。







