导读:测序市场竞争激烈,同时,测序公司、研究机构在测序技术研发上也你追我赶。目前,最受瞩目的当属纳米孔测序技术……
纳米孔技术读取DNA碱基(图:J. Gundlach/U. of Washington)
近年来,激烈的竞争使得DNA测序技术迅猛发展。现在,一种叫做纳米孔测序的技术犹如一匹黑马闯入大家的视线,看起来很有潜力在竞争中拔得头筹。研究人员首次证明,他们可以在DNA链通过一个微小的孔时连续地读出DNA的各个“化学字母”,这为研发解码DNA就如同播音员阅读自动收报机纸条般的测序仪铺平了道路。这种先进的技术有可能将完整测出人类基因组的费用降低到1000美元以下,为个体化医疗带来革命,并将基于遗传学的诊疗及医学带入新的时代。
大多数的测序工作需要耗时数天。测序仪对DNA双链进行复制,并利用荧光标记及其他成分进行修饰,从而对DNA链的碱基组成进行读取。纳米孔测序技术则无需这些额外的步骤,直接对未经修饰的DNA链进行读取,从而有可能成为市面上最快、最便宜的测序技术。
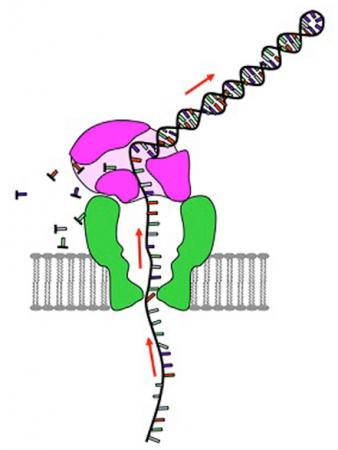
这个想法—使DNA链通过小孔并然后读取其碱基—最初是在1996年由麻省及加州的研究人员提出来的。后来,科学家们解决了如何利用电荷使DNA通过嵌入在在薄膜上的带小孔蛋白质。当DNA碱基通过孔时,它们使电荷发生变化。灵敏的电子设备检测到这些变变化从而鉴定所通过的碱基。
不过,存在的一个主要的问题是,当薄膜施加电压后,DNA通过纳米孔的速度过快而难以对序列上的碱基进行读取。两年前,加州大学的Mark Akeson和同事们偶然发现了一个可能的解决方案。他们将一种叫做phi29的蛋白质添加到纳米孔装置中。这种蛋白质在DNA运动通过纳米孔时可与DNA形成松散的结合,从而使DNA的运动速度慢了下来。
最近,华盛顿大学的物理学家Jens Gundlach所领导的一个研究团队在Nature Biotechnology上报道称,他们将Akeson的phi29蛋白整合至他们的纳米孔设备,这种纳米孔设备使用了一种不同的孔蛋白,可更快速地鉴定四种碱基。Phi29蛋白使DNA速度下降,使得每秒通过纳米孔的核酸碱基数为20至30个,从而有可能对每一个经过纳米孔的碱基进行识别。“这是纳米孔测序的独门绝技” Gundlach说。
该技术进步将加剧与另外一家叫做Oxford Nanopore Technologies公司的纳米孔测序技术的竞争。在2月份,Oxford Nanopore Technologies公司宣称,他们可以对通过一个单独纳米孔的DNA链进行完整的测序,并且有可能在2013年早期发售可同时运行数千个纳米孔的测序仪,从而有可能将全基因组测序时间缩短到15分钟,费用降到1000美元左右。
大多数的科学家认为,如果消息不假,这个技术则确实是个了不起的进步。不过他们仍旧在等待证明。“Oxford Nanopore首先发出了宣告,但是由于没有公布足够数据而受到严厉批评,”圣地亚哥的Electronic BioSciences公司总裁、化学家Geoffrey Barrall说。相反,Barrall说,Gundlach和同事们的成果看起来很有说服力。“这是第一篇真正进行了DNA测序的文章。”

 Reading DNA at single-nucleotide resolution with a mutant MspA nanopore and phi29 DNA polymerase
Reading DNA at single-nucleotide resolution with a mutant MspA nanopore and phi29 DNA polymerase
Elizabeth A Manrao, Ian M Derrington, Andrew H Laszlo, Kyle W Langford, Matthew K Hopper, Nathaniel Gillgren, Mikhail Pavlenok, Michael Niederweis & Jens H Gundlach
Nanopore technologies are being developed for fast and direct sequencing of single DNA molecules through detection of ionic current modulations as DNA passes through a pore's constriction. Here we demonstrate the ability to resolve changes in current that correspond to a known DNA sequence by combining the high sensitivity of a mutated form of the protein pore Mycobacterium smegmatis porin A (MspA)3 with phi29 DNA polymerase (DNAP)4, which controls the rate of DNA translocation through the pore. As phi29 DNAP synthesizes DNA and functions like a motor to pull a single-stranded template through MspA, we observe well-resolved and reproducible ionic current levels with median durations of ~28 ms and ionic current differences of up to 40 pA. Using six different DNA sequences with readable regions 42–53 nucleotides long, we record current traces that map to the known DNA sequences. With single-nucleotide resolution and DNA translocation control, this system integrates solutions to two long-standing hurdles to nanopore sequencing.
文献链接:https://www.nature.com/nbt/journal/vaop/ncurrent/full/nbt.2171.html







