虽然现在随着手机的普及和网络的发达,已经很少出现手机/手表时间不准的情况。不过如果发生这种情况,通常都需要将手机/手表与一个公认的标准时间进行同步。人类基因组计划的成果就是为科学家们提供了这样一份公认的“标准”——参考基因组。就像一个坐标系,研究人员可以对照参考基因组,更好地理解人类基因组的结构、功能和变异。
人类基因组计划于1990年开始,2001年完成工作草稿并发表,2003年完成最终测序图谱,堪称是人类生命科学史上里程碑式的成就。不过,这份“地图”并不是完整的,还留下了8%的空白。

图1 人类基因组计划LOGO(图源:维基百科)
这8%的空白主要是基因组中的异染色质区域。所谓异染色质,是相对常染色质而言。常染色质会被碱性染料染成浅色,或对福尔根反应呈弱阳性;异染色质则会被染成深色,或对福尔根反应呈阳性。异染色质或是在整个细胞周期内都处于凝聚状态,比如着丝粒、端粒、核仁形成区等部位;或是在一定的发育时期和生理条件下,由常染色质凝聚成异染色质,比如雌性哺乳动物的一对X染色体中,有一条会在胚胎发育得第16-18天凝聚为相对失活的巴氏小体。
异染色质高度凝聚,同时充满着重复序列,这就使得测序困难,也不好对测序结果进行组装。这是由于当时的技术仅能实现短读长测序,即测序的时候需要把基因组打断成较短的DNA片段,在对每个片段完成测序后,再通过一定的算法比对和匹配重叠序列,逐步将片段拼接成完整的基因组序列。因此,重复序列的存在极大地阻碍了片段的拼接和组装,而短读长测序意味着测序的DNA片段必须足够短,就像使用极小的碎片进行拼图,进一步增加了重建基因组的难度。
不过,随着测序技术的进步,人们能拿到的“碎片”越来越大,“拼图”的难度有所降低。2022年3月底,“端粒到端粒(Telomere-to-Telomere,T2T)”联盟公布了一个完整的人类基因组序列T2T-CHM13,补上了8%的空缺。该基因组序列源自一枚完全性葡萄胎(Complete Hydatidiform Mole, CHM),即只含有父源性基因组,而不含有母源性基因组。这使得重建基因组时,不必面对父源、母源基因组错误交叉组装的难题,但也使得最终的结果未能包含Y染色体的序列。
不过,T2T-CHM13和目前主流使用的人类参考基因组GRCh38(Genome Reference Consortium Human Build 38)一样,主要基于单个基因组。GRCh38是基因组参考联盟(Genome Reference Consortium,GRC)在人类基因组计划的基础上不断更新和维护,于2017年发布的最新版本,2022年3月发布了最新的补丁GRCh38.p14。该基因组的来源中,大约70%来自于纽约布法罗市的一名男性,23%来自于另外10人,剩下7%由超过50人贡献。据分析,该男性具有非洲-欧洲混合血统。
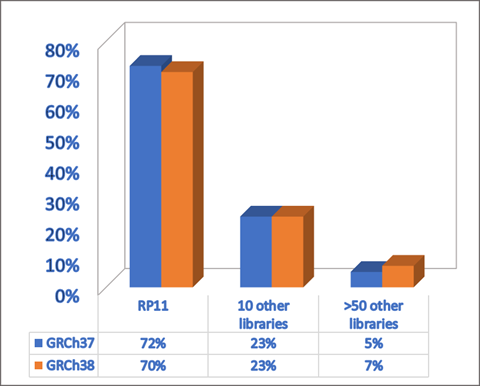
图2 不同基因组来源对GRCh38的贡献(图源:[3])
尽管地球上任何两个人基因组的相似性高达99.5%-99.9%,但显然,这不到1%的差异造就了人类的遗传多样性。而且,在涉及结构变异(Structural variation,SV)时,即基因组中发生较大片段的插入、删除、倒位或重复时,参考基因组无法正确地描述和表征。此外,还有数百兆碱基仅在一部分具有特定祖源的人群身上出现,并未被包含到参考基因组中。因此,仅使用单一的基因组作为参照是远远不够的。比如,在预测疾病风险时,对于黑人来说,仅对照现有的参考组,可能会产生较大的低估。
5月10日,由人类泛基因组参考联盟(Human Pangenome Reference Consortium,HPRC)制作的首个人类泛基因组参考草图在Nature上发布。与以往的参考基因组不同,这次的泛基因组草图包含了来自全球的47名具有不同祖先的个体的共计94份基因组,并对这47人的父母也进行了基因组的收集,以分析他们基因的父、母源性。

图3 泛基因组参考草图发布(图源:[4])
这47人包括来自非洲、美洲和亚洲的个体,其中也有来自中国的代表。其中许多样本来自于千人基因组计划(1000 Genomes Project),并由人类泛基因组联盟使用“长读长测序”的新技术进行重新分析。团队计划到2024年年中,将样本范围扩大到350人。
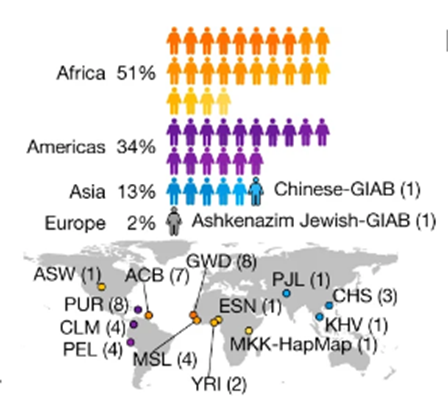
图4 47人来源(图源:[4])
相较于GRCh38,这次的结果在原先32亿碱基对的基础上新增了1.19亿个碱基对,其中,大约由9000万个来自结构变异。另外,团队还发现了 1115 个与进化有关的新基因重复。
为了更好地呈现泛基因组参考草图,纽约西奈山伊坎医学院的遗传学家Eimear Kenny和她的同事通过计算,将47人的序列排列到一起,绘制出一个和地铁线路图有些相似的“泛基因组图”。
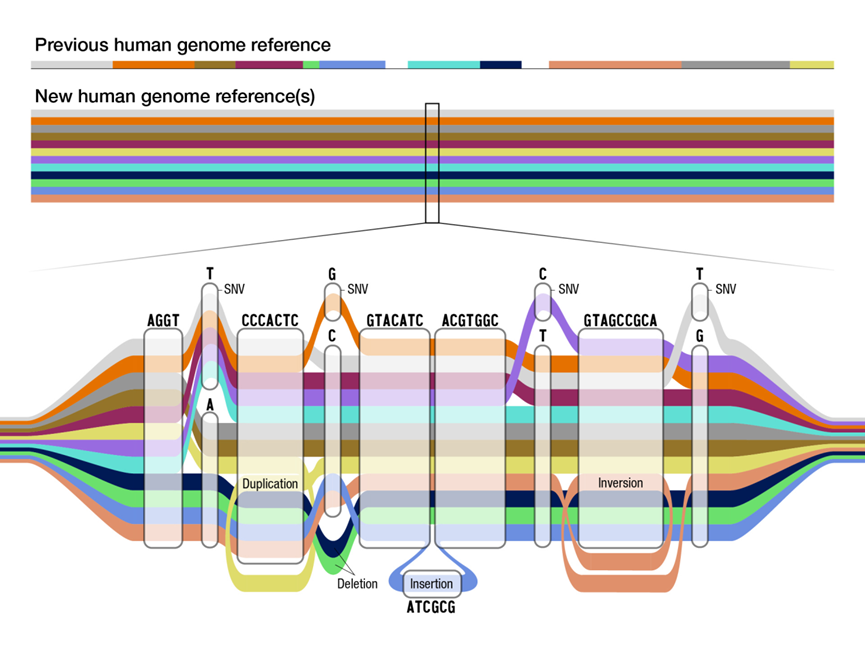
图5泛基因组参考(图源:国家人类基因组研究所)
在这张图上,碱基相同的部分与以往的单一基因组参考一样保持了线性,而在某些单个核苷酸差异的地方,出现了路线的分歧,另外,绕过某个核苷酸代表缺失(如上图绿色和深蓝色路径),顺时针环绕代表重复(如黄色路径),逆时针环绕代表倒位(如粉色路径)等等。
这次的泛基因组参考草图实现了预期序列99%的覆盖,并且在结构和碱基对水平上也达到了超过99%的准确度。与基于GRCh38 的工作流程相比,使用该草图分析短读长数据可降低34%的小遗传变异发现错误,并在检测单倍型结构变异时检出率增加了104%。
论文的第一作者、耶鲁大学的研究附属机构和圣路易斯华盛顿大学博士生Wen-Wei Liao表示:“人类泛基因组参考使得我们能够表征数以万计的新基因组变异,这些变异在以前是无法获得的。有了泛基因组参考,我们可以提高对基因和疾病之间联系的理解,从而加速临床研究。”
国家人类基因组研究所(National Human Genome Research Institute,NHGRI)主任医学博士 Eric Green表示:“基础研究人员或者临床医生,都应该具备接触到能够反映人口多样性的参考序列的机会,这样才能减少人类健康上的不公平性。人类泛基因组的创建和更新符合NHGRI在基因组学研究的各个方面上追求全球多样性的一贯目标,有助于推进基因组知识的传播,促进基因组医学以更公平的方式实施。”
目前,大多数参与HPRC的机构都位于美国和欧洲。HPRC成员机构、加利福尼亚大学圣克鲁兹分校的遗传学家Karen Miga表示,下一阶段该项目将向一项真正的国际间合作发展,并对历史上代表性不足的地区进行充分采样和测序。除了使用多样的基因组外,该项目还将积极接触样本人群并了解他们的医疗保健需求,使得这些人群能够直接从该项目的成果中收益。
参考资料:
[1]首个完整人类基因组图谱公布,长江日报记者对话6名中外专家
https://baijiahao.baidu.com/s?id=1730855664140430887&wfr=spider&for=pc
[2]Nurk S, Koren S, Rhie A, et al. The complete sequence of a human genome.Science376,44-53(2022).DOI:10.1126/science.abj6987
[3]https://www.ncbi.nlm.nih.gov/grc/help/faq/#human-reference-genome-individuals
[4]Liao, WW., Asri, M., Ebler, J. et al. A draft human pangenome reference. Nature 617, 312–324 (2023). https://doi.org/10.1038/s41586-023-05896-x
[5]Arya Massarat, Melissa Gymrek. (2023) A collective human reference genome. Nature
[6]https://www.genome.gov/news/news-release/scientists-release-a-new-human-pangenome-reference
[7]https://www.science.org/content/article/pangenome-hopes-represent-more-diverse-view-humans





