本文转载自“丁香学术”微信公众号。
遗传变异特征的解析和遗传变异谱的构建是研究人口、人口历史、医学遗传学和基因型-表型关联的基础。
自 2003 年第一次人类基因组信息发布以来,许多大规模的全基因组测序(WGS) 项目率先在西方国家启动,生成了大量和多样化的人口遗传变异资源。这是因为从大队列 WGS 资源构建单倍型参考面板是促进全基因组关联研究(GWASs)的一种有意义且成本效益高的方法。然而,直到目前,作为世界上最大的民族,中国汉族人群仍然没有具体的参考标准。
尽管前期的一些研究也关注过中国人口,但这些研究要么是样本量有限,要么是地理覆盖范围不够广,或者是基因组覆盖深度较浅,这些都限制了其作为参考的可信度。
在 2020 年针对中国人的一项研究中,ChinaMAP 项目提供了超过 1 万人的高深度(40X)全基因组测序数据和表型的系统性分析,但是该项目聚焦代谢性疾病,并且他们并未根据该研究构建单倍型参考面板。汉族人口是东亚乃至世界上最大的民族,约占全球人口的 20%,占中国大陆人口的 92%。因此,建立完整的、高质量的汉族群体遗传变异数据库和参考图谱势在必行,这样一种资源将有助于解析人口结构和人口历史,并促进世界上最大人口的遗传研究。
2021 年 11 月 16 日,中国科学院生物物理研究所徐涛院士团队和何顺民研究员团队合作在 Cell Reports 发表了题为 NyuWa Genome resource: A deep whole-genome sequencing-based variation profile and reference panel for the Chinese population 的文章,系统介绍了一种被称之为 NyuWa(女娲)的基因组资源,数据来自于 23 个中国省份中的约 3000 人的深度 WGS。
该数据资源是一个高质量的公开可用的中国人口特定的参考信息,目前在国际上具有最佳的汉族人口相关研究的参考价值,也为中国人群的遗传和疾病研究提供有用和可靠的支持。

(图片来源:Cell Reports)
主要研究内容
基于大型中国人口队列的深度 WGS 数据
NyuWa 基因组资源包括 2999 个不同中国样本的高测序深度的 WGS 数。样本来自中国 23 个行政区域,包括 17 个省、2 自治区和 4 个直辖市。大部分样本来自上海、广东和北京。经过基因组比对和去除重复后,实际基因组覆盖深度的中位数为 26.23。基于性染色体的基因组覆盖率,每个受试者的性别都可以被清楚地识别,共包括 1335 名女性和 1664 名男性。
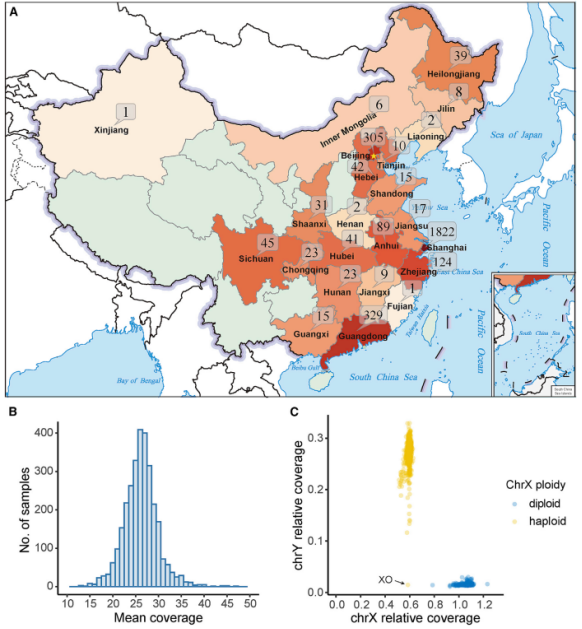
(图片来源:Cell Reports)
基于 NyuWa 数据发现了 2500 万个新变体
经过严格的质量控制,研究人员共鉴定出 7106 万 SNPs 和 819 万 InDels,并对其进行了全面注释。通过与其他变异信息公共数据库相比,NyuWa 数据集包含 2500 万个新的变异,包括 2310 万个 SNP(32.5%)和 190 万位点插入(23.3%)。进一步的分析发现,蛋白质编码基因共存在 3190 万个变异位点,其中 85.7 万个位于 CDS 区域,110 万个位于 UTR,3000 万个位于内含子。lncRNA 外显子区共有 478 万个变异。
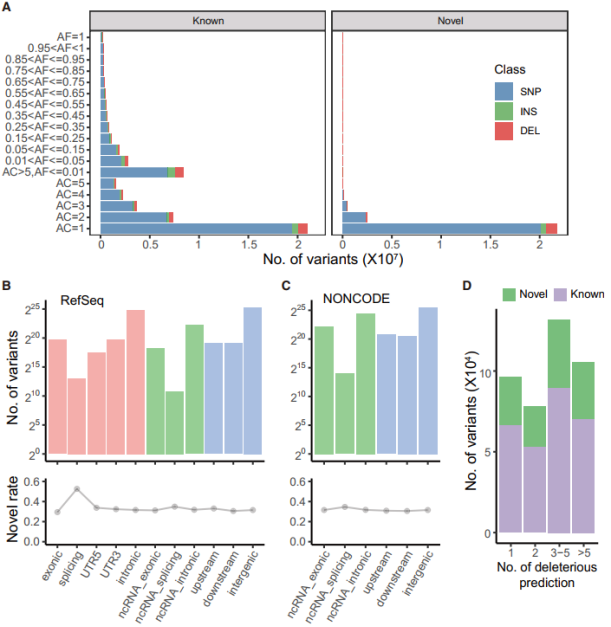
(图片来源:Cell Reports)
随后,为了评估 NyuWa 参考面板的基因型推演性能,研究团队使用来自人类基因组多样性计划(the Human Genome Diversity Project, HGDP)的亚洲各个人群芯片基因分型数据和高覆盖率 WGS 数据作为测试数据集。通过比较分析,他们发现 NyuWa 在对中国人口相关的基因组研究中优于其他现存的参考数据库。同时,NyuWa 在中国南方和北方人群中均适用。
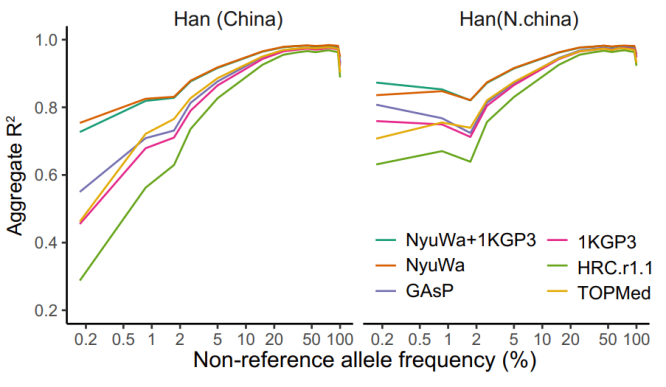
(图片来源:Cell Reports)
变体的临床价值
为了进一步说明 NyuWa 资源在改善人类健康方面的价值,他们进一步评价了其在基因突变相关疾病研究和医学应用方面的应用价值。正如预期的那样,NyuWa 和公共数据集中的大多数致病变异是一致的,均属于罕见变异。致病性变体通常频率很低,而那些等位基因频率较高的致病性变体可能与常见疾病有关。
此外,他们还发现了一些由 ClinVar 数据库对致病性的解释相互矛盾的变体,这些变体在 NyuWa 资源中显示出更高的等位基因频率。例如,以 1% 的等位基因频率作为阈值,两种变体 rs182677317 和 rs369849556 被注释为与罕见疾病睫状体运动障碍相关,而 NyuWa 数据集中的高等位基因频率(>1%)则表明这些变体可能不是致病性的。因此,这些结果表明,NyuWa 数据集中的变异等位基因频率可以为疾病相关变异的研究提供额外的参考。
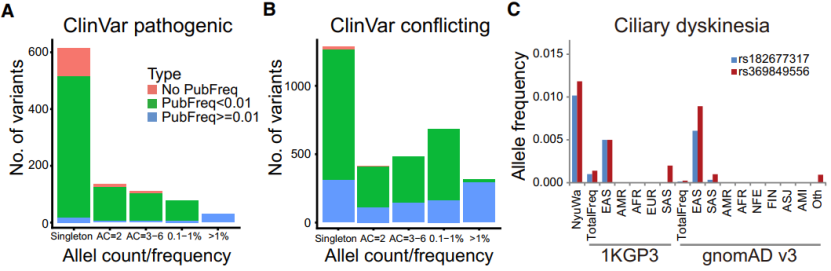
(图片来源:Cell Reports)
他们还评估了 ADME 核心基因的已知药物基因组位点的等位基因频率,而这可能影响中国不同省份和全球地区药物的疗效和安全性。结果发现一些变异在中国不同地区和世界不同人群中具有明显的等位基因频率差异。
例如,世界卫生组织推荐用于治疗结核病的药物异烟肼主要由 NAT2 酶(N-乙酰转移酶 2)乙酰化代谢,代谢的速率由遗传因素决定,代谢慢的患者发生肝毒性的风险更高。NAT2 存在多个 SNP 位点,形成不同的等位基因,其中一种称为 NAT2*12,为快乙酰化等位基因。研究人员发现在中国不同省份和东亚地区检测到 NAT2*12 的等位基因频率一致较高(接近 100%),而在其他人群中频率则较低。这表明中国人群在使用异烟肼前检测 NAT2*12 并不像其他人群那么必要。
而对于其他多种基因的检测,其等位基因的频率在中国不同省份之间存在差异,因此,建议在某些药物用于个体化治疗之前,进行基因检测。
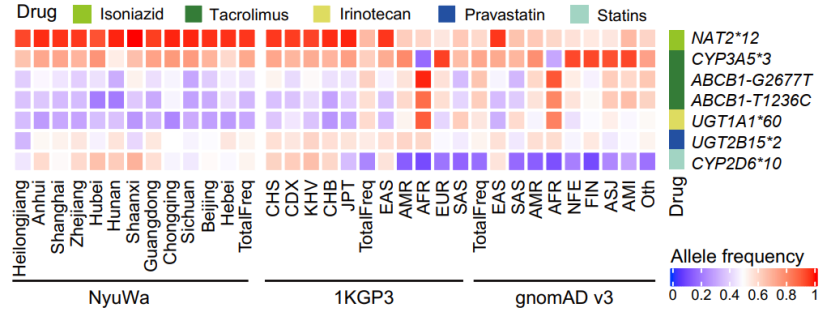
(图片来源:Cell Reports)
蛋白编码基因和 lncRNA 基因功能缺失的变异
功能缺失变异对基因功能有深刻的影响,并为临床基因组解释提供信息。在本研究中,他们筛选了高可信度的功能丧失型蛋白质截短变异体(Protein-Truncating Variants, PTVs),特别是那些新鉴定的变异体。他们在 7696 个基因中发现了 18711 个 PTVs。
进一步分析发现在 NyuWa 数据集中共鉴定了 9994 个新的 PTVs,其中纯合子数目为 21 个。另外,在 906 个癌症相关基因中的 385 个中检测到 1138 个 PTVs,其中 636 个为新型 PTVs。比如说,在 BRCA2、BRCA1、PMS1、TP53 和 MSH6 中鉴定了 5 种新型 PTVs 和 48 种已知 PTVs。
由于 lncRNA 不包含 CDS 区域,因此剪接变异成为 lncRNA 功能缺失变异中最重要的一类。剪接变异可能导致内含子保留或外显子跳跃,从而极大地改变了 lncRNA 的序列和结构。NyuWa 数据集共发现 3544 个 lncRNA 基因中的 3793 个剪接变异。包括 Ensembl 数据库中 1287 个 lncRNA 基因的 1454 个剪接变异和 NONCODE 数据库中 2257 个 lncRNA 基因的 2339 个剪接变异。据报道,230 个 lncRNA 基因对细胞生长至关重要,他们也在其中 20 个 lncRNA 基因中发现了 22 个剪接变体,等位基因频率大于 0.1% 的 lncRNA 剪接变异所占比例小于所有的 lncRNA 剪接变异,表明剪接变异体可以真正影响这些 lncRNAs 的功能。
因此,NyuWa 数据集中发现的蛋白编码和非编码基因的功能缺失变异可能与疾病的病因学或遗传倾向有关,这将为疾病和遗传学研究提供新的见解。
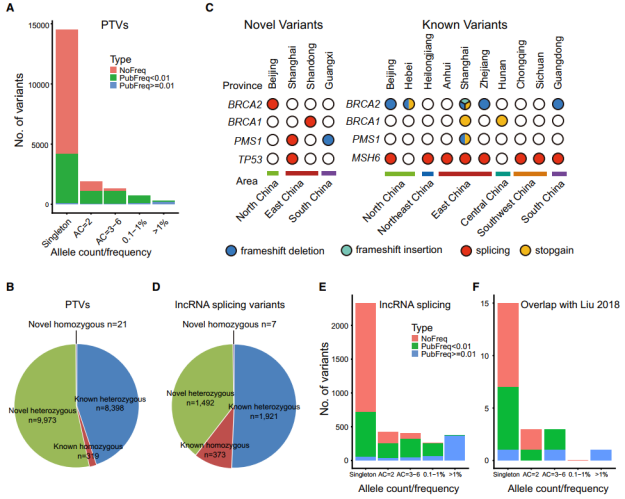
(图片来源:Cell Reports)
结语
综上所述,在这项研究中,他们报道并描述了 NyuWa 资源库,其包括中国人口的大队列深度 WGS 数据,并基于此构建了中国人群的遗传变异图谱和首个数千人级别公开可用的中国人群单倍型参考面板。
基于 NyuWa 数据发现的 2500 万个新变异,将极大地推动人类疾病的研究,特别是中国人群的疾病研究;这项工作的另一个重要贡献是可以填补基于 WGS 的中国人口单体型参考面板的空白。
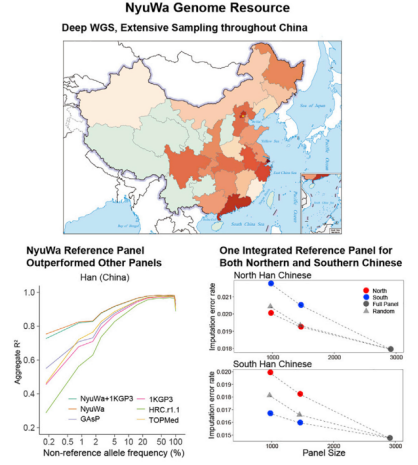
(图片来源:Cell Reports )
同时,本研究中对蛋白质编码基因和 lncRNA 基因的功能缺失变异体的鉴定扩展了目前已知的功能缺失变异体的目录,当与表型信息相结合时,该资源将为功能基因组学研究提供重要的参考。
有许多因素会影响我们的睡眠质量以及我们是否经历认知衰退。虽然有些因素是不可预防的,如:遗传易感性,但除了睡个好觉之外,我们还可以做很多事情来降低患痴呆症的可能性,例如:锻炼身体和健康饮食。
参考资料:
1.Zhang et al., NyuWa Genome resource: A deep whole-genome sequencing-based variation profile and reference panel for the Chinese population, Cell Reports (2021).
2.Cao, Y., et al.; ChinaMAP Consortium (2020). The ChinaMAP analytics of deep whole genome sequences in 10,588 individuals. Cell Res.30, 717–731. [3]Sud, A., Kinnersley, B., and Houlston, R.S. (2017). Genome-wide association studies of cancer: current insights and future perspectives. Nat. Rev. Cancer 17, 692–704.